プレスリリース

生体内でのひとつひとつの細胞におけるRNA分子を正確に数えることに成功
~生命動態や疾患動態の⌈ゆらぎ⌋へメスを入れる新たな技術の開発~
~生命動態や疾患動態の⌈ゆらぎ⌋へメスを入れる新たな技術の開発~
2015年2月26日
株式会社 国際電気通信基礎技術研究所(ATR)
株式会社 国際電気通信基礎技術研究所(ATR)
【概要】
株式会社国際電気通信基礎技術研究所(以下⌈ATR⌋、本社:京都府相楽郡精華町、代表取締役社長 平田康夫)社会メディア総合研究所·佐藤匠徳特別研究所の佐藤匠徳所長及び岡祐馬研修研究員は、脊椎動物の形態形成、またヒト疾患モデルの代表的な動物モデルであるゼブラフィッシュにおいて、一細胞レベルでRNA分子を数えることのできる一分子蛍光in situハイブリダイゼーション(single-molecule fluorescence in situ hybridization, smFISH)法の確立に成功しました。今回の研究により、ゼブラフィッシュ胚において一細胞レベルでmRNAの数を数えることが可能になり、脊椎動物における遺伝子発現の細胞間のばらつきとその役割に関する研究の進展が期待されます。 この成果は、Scientific Reports誌(電子版:英国時間2015年2月25日AM10:00公開)に掲載されました。
【背景】
遺伝子はDNAからmRNAに転写され、さらにタンパク質に翻訳されることで情報が読み取られ機能を発揮します。この過程を遺伝子発現と呼びます。遺伝子発現は確率論的な反応であるため、遺伝的に同一の細胞であっても、細胞間では遺伝子発現量にばらつきがあることが知られています。このばらつきを定量するためには一細胞レベルでのmRNA分子の数を数えることが必要です。これまで、一細胞レベルでmRNA量を定量する方法としてsmFISH法、一細胞リアルタイムPCR法および一細胞RNAシーケンス法という方法が開発されてきました。しかし、脊椎動物において、個体内の細胞の位置情報を保ったまま遺伝子発現量を定量する方法はありませんでした。
【研究内容】
smFISH法は細胞を破壊する必要がないのが特徴であり、細胞の構造を保ったままmRNA分子の数を数えることができます。しかし、これまでsmFISHは単細胞生物や培養細胞、無脊椎動物における方法が主に開発されており、脊椎動物では組織切片に対する方法しか存在しませんでした。そこで本研究では、脊椎動物モデルとして様々な研究で使われるゼブラフィッシュ胚を、切片を調整することなく胚をそのまま使用したsmFISH法の確立に取り組みました。
本研究では培養細胞におけるsmFISH法を基に条件検討を行いました。smFISH法では、細胞を固定した後に検出用の核酸分子をmRNAにハイブリダイズさせます。条件検討の結果、核酸分子のハイブリダイゼーションの前にメタノール処理をすることが重要だということがわかりました。メタノール処理はゼブラフィッシュ胚における一般的なin situハイブリダイゼーションでも行われる処理で、脱水作用があります。
メタノール処理の追加により、遺伝子特異的なmRNAの数を数えることに成功しました(図1)。また、二種類の検出用の核酸分子を同時に用いることで同一細胞において2種類のmRNAの数を同時に数えることにも成功しました。
【今後の展望】
今回の研究により、ゼブラフィッシュ胚において一細胞レベルでmRNAの数を数えることが可能になり、脊椎動物における遺伝子発現の細胞間のばらつきとその役割に関する研究が進展します。他の一細胞解析技術と組み合わせることで、生物の発生や病気の進行など様々な生命現象にどう遺伝子発現の細胞間のばらつきが関わっているか詳細に解析することができ、新たな側面からの生命現象のメカニズムの解明につながります。これらの研究の進展は、将来、今までになかった新しい病気の診断や治療薬の開発に貢献することが期待できます。
【論文情報】
Scientific Reports誌(電子版:英国時間2015年2月25日AM10:00公開)
Yuma Oka & Thomas N.Sato Whole-mount single molecule FISH method for zebrafish embryo
【用語解説】
- RNA:リボ核酸。DNAから遺伝情報がRNAに転写され、その情報を基にタンパク質が合成される。
- in situハイブリダイゼーション:組織や細胞において、DNAやRNAを核酸分子のプローブを使って検出する方法。DNAやRNAを抽出する必要がなく、組織や細胞の形状を保った状態で検出できる。
- リアルタイムPCR法:ポリメラーゼ連鎖反応(PCR)により鋳型のDNAは一定の増幅率で増幅される。この増幅を経時的に測定することで、増幅率に基づいて鋳型のDNAの定量を行う方法。
- RNAシーケンス法:組織中や細胞中に存在するRNAの配列を読み取ることで、組織中や細胞の遺伝子発現量を網羅的に定量する方法。
【研究グループ】
本研究はATR社会メディア総合研究所·佐藤匠徳特別研究所の岡祐馬研修研究員(おかゆうま;京都大学大学院 ·博士後期課程、JST ERATO佐藤ライブ予測制御プロジェクト)とATR社会メディア総合研究所·佐藤匠徳特別研究所の佐藤匠徳特別研究所長(さとうなるとく;JST ERATO佐藤ライブ予測制御プロジェクト研究総括、米国コーネル大学教授、豪州センテナリー研究所教授)の共同研究成果です。
※( ) 内は氏名、よみ、勤務先を表記。
【研究サポート】
本研究は、科学技術振興機構戦略的創造研究推進事業(ERATO)、日本学術振興会科研費基盤S(22229007)および武田科学基金の助成を受け実施されました。
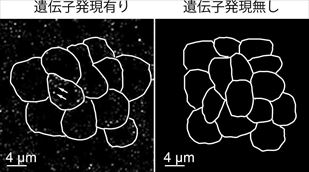
図1. 受精後12時間目のゼブラフィッシュ胚に対するsmFISHの結果。一つのドット状のシグナル(矢印)がひとつひとつのmRNA分子を示す。遺伝子発現がある細胞のみにドット状のシグナルが得られた。白線は細胞境界を表す。

